from sklearn import cluster, datasets
# 讀入鳶尾花資料
iris = datasets.load_iris()
iris_X = iris.data
# KMeans 演算法
kmeans_fit = cluster.KMeans(n_clusters=3).fit(iris_X)
# 印出分群結果
cluster_labels = kmeans_fit.labels_
print("分群結果:")
print(cluster_labels)
print("---")
# 印出品種看看
iris_y = iris.target
print("真實品種:")
print(iris_y)
一種密度基礎的空間分群方法,用於將資料點分成集群(cluster)和噪聲點(noise)
資料點(Data Points)
資料集中的個體或觀測值,通常表示為N維空間中的點。
半徑(Radius,ε)
DBSCAN使用半徑來定義每個資料點的鄰域範圍,資料點在這個範圍內視為鄰居。
核心點(Core Point)
核心點是指在其半徑範圍內擁有至少MinPts個鄰居的資料點。
邊界點(Border Point)
邊界點是指在其半徑範圍內鄰居數量不足MinPts的資料點,但可以連接到一個核心點而成為該集群的一部分。
噪聲點(Noise Point)
噪聲點是指在半徑範圍內無核心點鄰居的資料點,它們被視為孤立的資料點或雜訊。
ε-鄰域(ε-Neighborhood)
ε-鄰域表示以某個資料點為中心,半徑為ε的區域,包含在這個區域內的所有資料點被視為鄰居。
核心物件集合(Core Object Set)
核心物件集合包含所有核心點及其相關的連接邊界點。
邊界物件集合(Border Object Set)
邊界物件集合包含所有邊界點,這些點是與核心點相關聯的資料點。
import matplotlib.pyplot as plt
from sklearn.cluster import DBSCAN
import numpy as np
data = np.array([[1, 2], [5, 8], [1.5, 1.8], [8, 8], [1, 0.6], [9, 11]])
# 初始化DBSCAN模型,指定eps(半徑)和min_samples(最小樣本數)
dbscan = DBSCAN(eps=2, min_samples=2)
# 適配模型到數據
dbscan.fit(data)
# 獲取每個數據點的分群標籤,-1 表示噪聲點
labels = dbscan.labels_
print("分群標籤:", labels)
# 取得 DBSCAN 的分群結果
core_samples_mask = np.zeros_like(labels, dtype=bool)
core_samples_mask[dbscan.core_sample_indices_] = True
# 繪製分群結果
unique_labels = set(labels)
colors = [plt.cm.Spectral(each)
for each in np.linspace(0, 1, len(unique_labels))]
for k, col in zip(unique_labels, colors):
if k == -1:
# 噪聲點以黑色表示
col = [0, 0, 0, 1]
class_member_mask = (labels == k)
xy = data[class_member_mask & core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col),
markeredgecolor='k', markersize=14, label=f'Cluster {k}')
xy = data[class_member_mask & ~core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col),
markeredgecolor='k', markersize=6)
plt.title('DBSCAN Clustering Result')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.legend(loc='best')
plt.show()
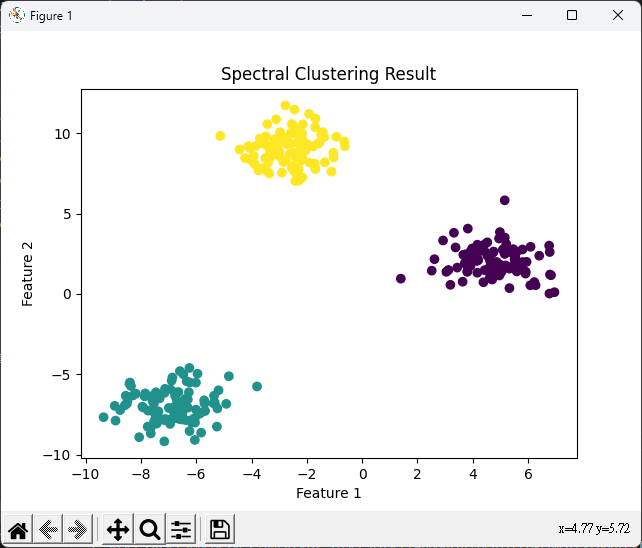
譜分群是一種基於資料的特徵間的相似性矩陣進行分群的方法,通常用於圖分割和文本分類等應用。
import numpy as np
from sklearn.cluster import SpectralClustering
from sklearn.datasets import make_blobs
import matplotlib.pyplot as plt
data, ground_truth = make_blobs(
n_samples=300, centers=3, cluster_std=1.0, random_state=42)
# 初始化譜分群模型,指定集群數和相似性矩陣的配置
n_clusters = 3
spectral_clustering = SpectralClustering(
n_clusters=n_clusters, affinity='nearest_neighbors')
# 適配模型到數據
labels = spectral_clustering.fit_predict(data)
# 繪製分群結果
plt.scatter(data[:, 0], data[:, 1], c=labels, cmap='viridis')
plt.title('Spectral Clustering Result')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.show()
from sklearn.metrics import homogeneity_score, completeness_score, adjusted_rand_score
import numpy as np
# 真實分群結果
true_labels = np.array([0, 0, 1, 1, 2, 2])
# 假設的分群結果
predicted_labels = np.array([0, 0, 2, 2, 1, 1])
# 計算同質性
homogeneity = homogeneity_score(true_labels, predicted_labels)
# 計算完整性
completeness = completeness_score(true_labels, predicted_labels)
# 計算ARI
ari = adjusted_rand_score(true_labels, predicted_labels)
print("同質性:", homogeneity)
print("完整性:", completeness)
print("ARI:", ari)
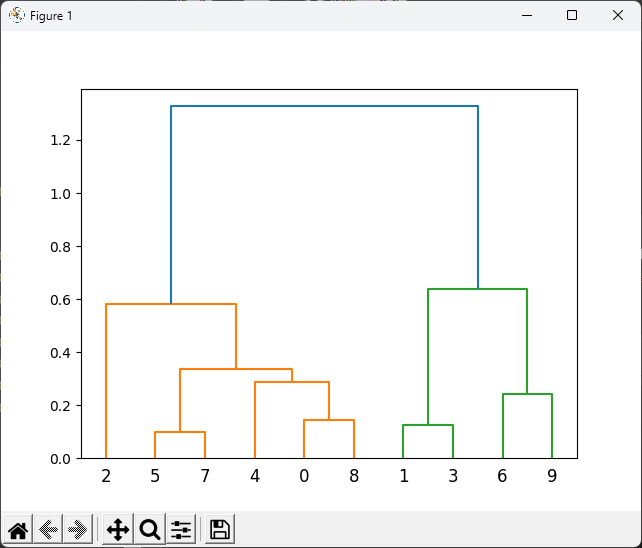
from scipy.cluster.hierarchy import linkage, dendrogram
import matplotlib.pyplot as plt
import numpy as np
data = np.random.rand(10, 2)
# 計算聚合連結矩陣
linkage_matrix = linkage(data, method='ward')
# 繪製樹狀圖
dendrogram(linkage_matrix)
plt.show()
階層式分群中的相似性通常使用距離度量。
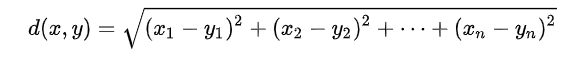
from scipy.spatial.distance import euclidean
point1 = [1, 2]
point2 = [4, 6]
distance = euclidean(point1, point2)
print("歐氏距離:", distance)
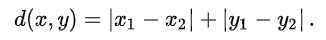
from scipy.spatial.distance import cityblock
point1 = [1, 2]
point2 = [4, 6]
distance = cityblock(point1, point2)
print("曼哈頓距離:", distance)
from sklearn.metrics.pairwise import cosine_similarity
import numpy as np
vector1 = np.array([1, 2, 3])
vector2 = np.array([4, 5, 6])
similarity = cosine_similarity([vector1], [vector2])
print("餘弦相似度:", similarity[0][0])
計算兩個集群之間的相似度。
from scipy.cluster.hierarchy import linkage, dendrogram
import matplotlib.pyplot as plt
data = np.array([[1, 2], [2, 3], [4, 5], [5, 6]])
# 使用完整連結進行階層分群
linkage_matrix = linkage(data, method='complete')
# 繪製階層分群樹
dendrogram(linkage_matrix)
plt.show()
from scipy.cluster.hierarchy import linkage, dendrogram
import matplotlib.pyplot as plt
data = np.array([[1, 2], [2, 3], [4, 5], [5, 6]])
# 使用平均連結進行階層分群
linkage_matrix = linkage(data, method='average')
# 繪製階層分群樹
dendrogram(linkage_matrix)
plt.show()
from scipy.cluster.hierarchy import linkage, dendrogram
import matplotlib.pyplot as plt
data = np.array([[1, 2], [2, 3], [4, 5], [5, 6]])
# 使用沃德連結進行階層分群
linkage_matrix = linkage(data, method='ward')
# 繪製階層分群樹
dendrogram(linkage_matrix)
plt.show()
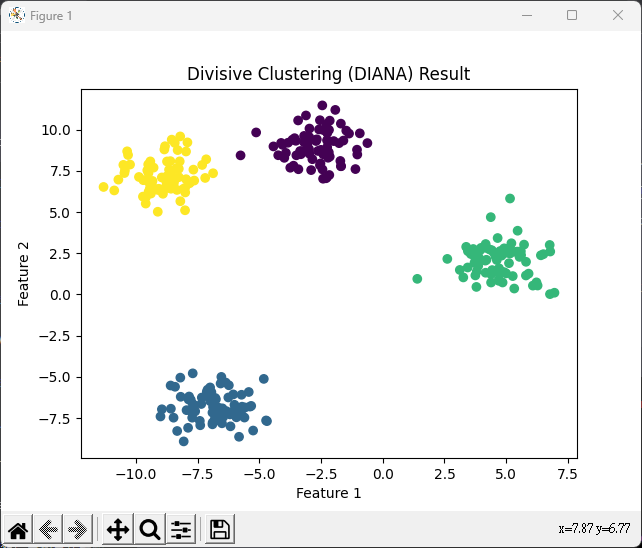
from sklearn.cluster import AgglomerativeClustering
from sklearn.datasets import make_blobs
import numpy as np
import matplotlib.pyplot as plt
data, ground_truth = make_blobs(
n_samples=300, centers=4, cluster_std=1.0, random_state=42)
# 初始化階層式分群模型,並指定要分成的子集群數
n_clusters = 4
diana = AgglomerativeClustering(
n_clusters=n_clusters, affinity='euclidean', linkage='ward')
# 適配模型到數據
diana.fit(data)
# 獲取每個數據點的分群標籤
labels = diana.labels_
# 繪製數據和分群結果
plt.scatter(data[:, 0], data[:, 1], c=labels, cmap='viridis')
plt.title('Divisive Clustering (DIANA) Result')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.show()